Mise en place de la Spatial Transcriptomic
Les plateformes Microdissection laser et Transcriptome (PUMA, Neurocentre Magendie) souhaitent développer la Spatial Transcriptomic.
En effet, Frédéric Martins, ingénieur récemment arrivé sur la plateforme Transcriptome, a déjà réalisé ce type d’expérience dans son unité précédente. Il recherche donc des utilisateurs intéressés.
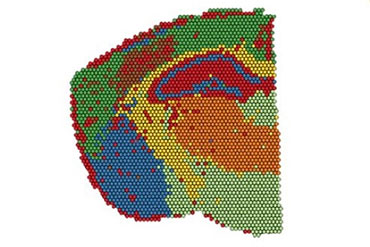
Le but de la Spatial Transcriptomic (technologie Visium, 10x Genomics) est de capturer les ARNm via leur queue poly-A et de les taguer avec un barcode spatial qui permet ensuite lors de l’analyse de retrouver la zone d’où provenait cet ARNm.
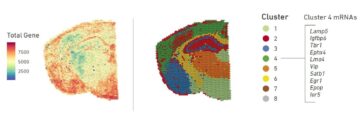 L’analyse du transcriptome se fait avec une résolution spatiale de 50µm et permet :
L’analyse du transcriptome se fait avec une résolution spatiale de 50µm et permet :
– d’évaluer l’expression différentielle entre zones de nos tissus (par ex. entre l’hippocampe, le thalamus et l’hypothalamus)
– de connaitre les gènes le plus exprimés par zone
– d’avoir la liste des gènes co-exprimés
– de représenter une cartographie gène à gène
Cette technologie nécessite pas d’investissement en termes d’équipement et permet de travailler sur des coupes de tissus congelées (tous organismes) ou FFPE (humain et souris) avec des tailles de 6,5mm x 6,5mm soit l’équivalent d’un hémisphère de souris. Cette technologie est donc parfaitement adaptée à l’étude du cerveau.
Contact
PUMA, Neurocentre Magendie
Frédéric Martins
05 57 57 36 79
Mise à jour: 16/02/23
