J.B. Sibarita, A. Beghin et al. dans Nature Methods
Localization-based super-resolution imaging meets high-content screening.
Beghin A, Kechkar A, Butler C, Levet F, Cabillic M, Rossier O, Giannone G, Galland R, Choquet D, Sibarita JB. Nat Methods. 2017 Oct 30. doi: 10.1038/nmeth.4486. [Epub ahead of print]
Jean-Baptiste Sibarita team leader et Anne Beghin: Quantitative Imaging of the Cell Institut Interdisciplinaire de Neurosciences / Bordeaux Neurocampus
La complexité immense du vivant requiert des approches systématiques à grande échelle pour trouver de nouvelles molécules qui pourraient être le point de départ de nouveaux médicaments. L’équipe de recherche « Imagerie Quantitative de la Cellule » menée par Jean-Baptiste Sibarita, en collaboration avec plusieurs autres équipes au sein de l’Institut Interdisciplinaire de Neurosciences de Bordeaux (IINS UMR CNRS 5297), a développé une nouvelle approche de microscopie de super-résolution basée sur la localisation de molécules uniques permettant le criblage haut-débit de conditions biologiques. Cette technique novatrice devrait permettre l’utilisation des méthodes de nanoscopie dans le processus de développement et d’identification de nouveaux agents thérapeutiques ciblant diverses pathologies.
Depuis plus d’une vingtaine d’années, Les laboratoires de biologie et l’industrie pharmaceutique utilisent quotidiennement des approches de microscopie dites de « criblage à haut débit », ou High Content Screening (HCS), afin de tester l’efficacité d’un grand nombre de molécules le plus rapidement possible. Plus récemment, le développement des approches de microscopie de super-résolution (SR) par localisation de molécules uniques permet de collecter un ensemble de nouveaux paramètres des propriétés cellulaires à l’échelle nanométrique. Il devient ainsi possible de décortiquer les propriétés d’organisation et de dynamique de millions de molécules par cellule.
Néanmoins, ces technologies étaient jusqu’à présent relativement lentes et manuelles et de fait totalement inadaptées au criblage selon les standards industriels ou ceux des laboratoires de recherche appliquée.
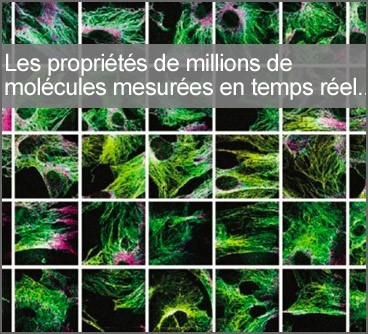
L’équipe interdisciplinaire composée de chercheurs et ingénieurs a réussi à développer une nouvelle approche demicroscopie de super-résolution basée sur la localisation de molécules uniques permettant le criblage haut-débit de centaines de conditions biologiques. Pour la première fois, il est possible de suivre au cœur des cellules vivantes l’effet direct de différents traitements pharmacologiques et/ou de conditions biologiques sur l’organisation et la dynamique de centaines de millions de molécules avec une précision nanométrique. Pour ce faire, les auteurs ont exploité leur savoir-faire unique dans le domaine de la microscopie de super-résolution pour développer un nouvel instrument révolutionnaire. Celui-ci est composé d’un microscope entièrement automatisé et d’une plateforme logicielle innovante permettant de piloter la séquence de criblage, de traiter en temps réel la quantité massive d’informations générée par la technique de super-résolution et de représenter ces données de manière intelligible pour toutes les conditionsbiologiques testées. Un prototype fonctionnel, facile d’utilisation par des équipes de recherche de différents domaines (neuroscience, cancérologie, immunologie…), est localisé au sein de l’IINS à Bordeaux. Il devrait notamment intégrer le processus de recherche des industries pharmaceutiques, à travers des collaborations d’ores et déjà prévues, et contribuer à identifier de nouveaux traitements en immunothérapie des cancers.
Exemple de criblage de super-résolution sur le cytosquelette de cellules cancéreuses réalisé sans intervention manuelle sur une plaque 96 puits, générant une base de données de plusieurs centaines de millions de données de molécules.
La description de la méthode a été publiée dans la revue Nature Methods, « When high content screening meets super resolution microscopy » par Beghin et al.
Ce développement a reçu le soutien de la Région Nouvelle Aquitaine, de l’infrastructure France Bio Imaging, du LabEx BRAIN et de l’IdEx de l’Université de Bordeaux.
Mise à jour: 19/06/18
